R语言编程入门(爬虫函数包的使用)
写在前面的
上期推文介绍了R语言编程入门,从了解R语言的5种数据结构和两种控制结构开始,再到借上上篇推文的Readscount数据综合利用R语言编程证明Readscount属于负二项分布。现在再回顾一下R语言的五个数据结构:向量、矩阵、列表、数据框、因子,以及两种控制结构,循环和判断。
今天我们来学习R语言编程的进阶——函数包的使用:掌握了一定R语言编程语法后,就要面对更高阶的学习了,我在前面说过,编程最后看的不是你对编程语言的掌握程度,而是看的你的数学能力和逻辑能力。当然在实际的生物信息学或者生物学研究生的工作中,我们没有那么多时间来进行数学模型建立、编程。那么巧妙应用已有的R语言函数包则是完成工作的捷径。R语言函数包非常多,积累并掌握一定数量的R语言函数包对于工作数据处理是有很显著的效率提升!为了让大家学会如何自学R语言函数包,我今天介绍一个大家听说过但又几乎没有接触过的内容——爬虫。
R语言爬虫
那什么是爬虫呢?爬虫又称网络爬虫,网络爬虫是一种按照一定的规则,自动地抓取互联网信息的程序或者脚本。也就是要通过一个程序将互联网网页上的内容获取,互联网上的内容是按照一定的格式或规范展示的,要通过程序获取互联网的内容就需要了解互联网的信息的格式。一般情况下互联网信息都使用一种叫HTML的格式,这种格式的定义是超级文本标记语言(英语:HyperText Markup Language,简称:HTML)是标准通用标记语言下的一个应用,也是一种规范,一种标准,它通过标记符号来标记要显示的网页中的各个部分。网页文件本身是一种文本文件,通过在文本文件中添加标记符,可以告诉浏览器如何显示其中的内容(如:文字如何处理,画面如何安排,图片如何显示等)。浏览器按顺序阅读网页文件,然后根据标记符解释和显示其标记的内容,对书写出错的标记将不指出其错误,且不停止其解释执行过程,编制者只能通过显示效果来分析出错原因和出错部位。但需要注意的是,对于不同的浏览器,对同一标记符可能会有不完全相同的解释,因而可能会有不同的显示效果。 “超文本”就是指页面内可以包含图片、链接,甚至音乐、程序等非文字元素。 超文本标记语言的结构包括“头”部分(英语:Head)、和“主体”部分(英语:Body),其中“头”部提供关于网页的信息,“主体”部分提供网页的具体内容。
简而言之,HTML是一种标记语言,通常情况下是由<元素>内容</元素>的形式存在,比如你新建一个txt文本文件在里面输入
Bioinformatic保存后修改.txt后缀为.html格式,再用你自己的浏览器打开就会出现下图效果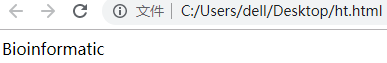
所以在每个HTML文件中,每个内容都会在某个或者某些个元素之下即<元素1><元素2><元素n>你的内容</元素1></元素2></元素3>,所以我们要找相关内容只需要找到这些内容对应的元素集合,我们将之称为xpath。所以我们只要找到目标信息对应的xpath就可以批量找到目标信息。读到这里可能很多读者也是蒙圈的,毕竟不是计算机专业出生不会这么快理解这些概念。但是不用担心,有一个Google浏览器插件SelectorGadget,这个可以自动帮助你抓取你想要的信息的路径。下面以一个实际例子来学会如何使用R语言爬虫的函数包。首先安装R语言爬虫函数包rvest
#安装rvest
install.packages(“rvest”)
其次在百度搜索SelectorGadget下载,拖拽到谷歌浏览器的拓展程序界面进行安装。
以PeerJ文献期刊数据库为爬虫网站,选择数据库的Life & Biology,出现以下内容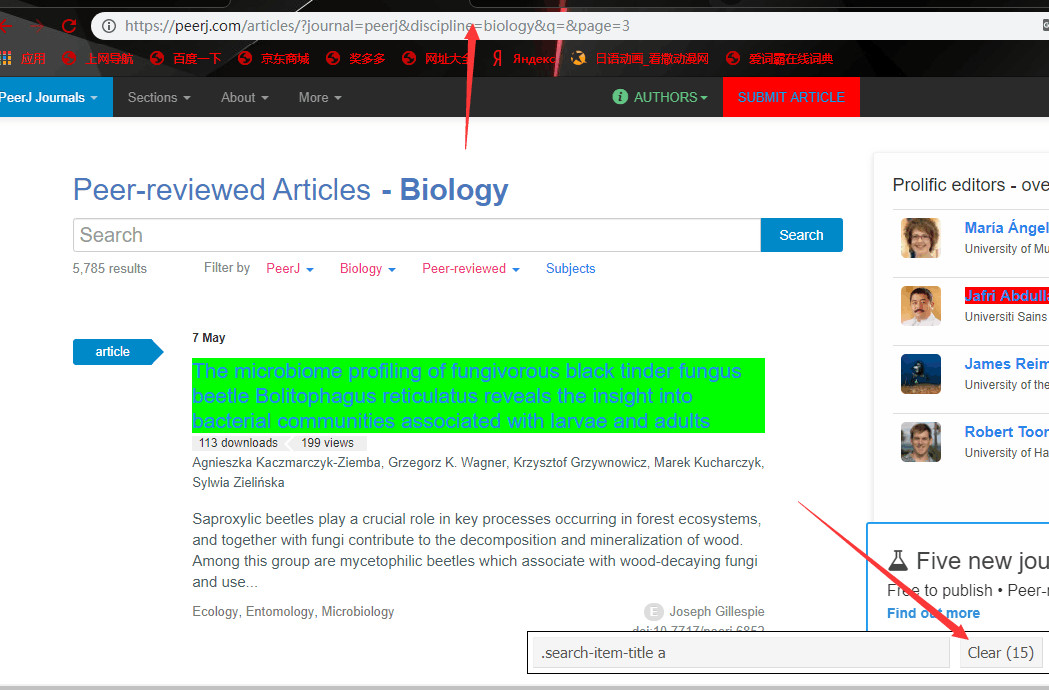
注意观察我的图片红色向上箭头指着的部分,这个是网页的变化地址代表第几页。所以在R语言里可以用一个循环来动态访问这里的每一页。接下来就是用SelectorGadget抓取每篇文章的注意观察上图中红色箭头标出的部分,由于网页中可能有其他版块的CSS与你想抓取的部分是一样的,要注意观察Clear中的数量是不是与你在当前页面想抓取的版块数量一致。比如我只想抓取每篇文章的题目,一页放了15篇文章的链接,所以数量是15个。如果数量大于你的预期数量应该找找有没有被多抓取的版块,再用鼠标点击一下他们就可以删除掉多余部分。接下来就是如何利用rvest包进行爬虫了。要学会一个新的函数包首先应该在R语言里使用??函数
??rvest
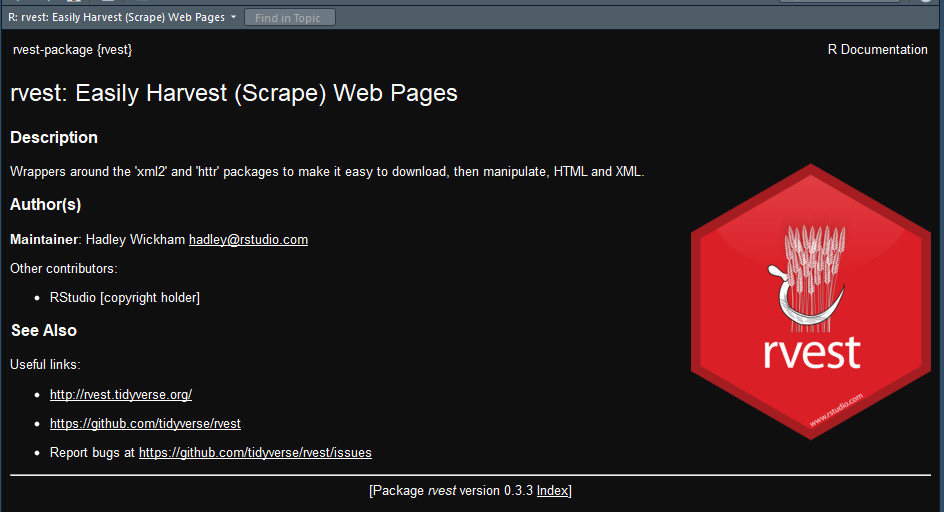
Rstudio的右下部分会弹出这个界面,里面说明了一些有用的教程网站。具体教程读者可以点击进去学习。这里我主要介绍三个有用的爬虫函数及管道符号的使用。首先爬虫的三个重要函数是read_html、html_nodes、html_text。这三个函数无需了解其余参数。首先read_html函数的意思是让R访问并读取你想爬虫的网页,html_nodes函数则是输入你想抓取的信息的xpath路径或者CSS路径(xpath和CSS指代相同的能找到该信息的路径,只是语法不同,例如用SelectorGadget抓取的是CSS路径,那么html_nodes函数中则输入css = ),html_text函数则是将爬虫到的信息以文本形式传递给变量。而管道在R语言中的作用则是将上一条命令的输出作为下一条命令的输入,下面的代码中将会利用管道符串联爬虫函数。
以爬取Peerj文献期刊新发表文献的题目,并可视化这些题目都有哪些词汇的例子来讲解何使用rvest函数包并强化R语言编程能力
#加载rvest函数包
library(rvest)
#先定义Bio_Paper变量用于储存爬虫PeerJ数据库中新发表的文献题目
Bio_Paper<-NULL
for (i in 1:7) {
#在循环中动态标记要爬虫网页的地址,由于所有网页的不同点在于page后的页码不同,所以使用paste函数在每次循环中粘贴不同的页码
url<-paste("https://peerj.com/articles/?journal=peerj&discipline=biology&q=&page=",i,sep = "")
#使用read_html函数读取网页信息
myPeerJ<-read_html(url)
#使用管道符将myPeerJ变量内容作为输入内容,并根据css路径用html_nodes抓取文献题目,又以管道操作符传递给html_text函数以文本方式传递回peerjBio
peerjBio<-myPeerJ %>%
html_nodes(css = ".search-item-title a") %>%
html_text()
#使用正则表达式函数将爬取的网页信息中的换行符替换掉,gsub函数中pattern参数是指代想被替换的内容,replacement函数是想替换的内容
peerjBio<-gsub(pattern = "\n",replacement = "",peerjBio)
#使用正则表达式函数将爬取的网页信息中多余空格替换掉
peerjBio<-gsub(pattern = " ",replacement = "",peerjBio)
#依次将peerjBio中的信息传递给Bio_Paper
Bio_Paper<-c(Bio_Paper,peerjBio)
}
#先定义一个列表类型变量,用于最后储存文献题目所用的词汇信息和词频信息
Bio_list<-list()
使用While循环统计词汇和词频信息,这里是一个重点,希望读者认真思考一下这里的代码为什么是这样写的(提示:列表的标签具有唯一性!)
i<-1
while (i<=length(Bio_Paper)) {
word<-strsplit(as.character(Bio_Paper[i])," ")[[1]]
for (j in 1:length(word)) {
wd<-word[j]
Bio_list[[wd]]<-c(Bio_list[[wd]],j)
}
i=i+1
}
#将列表的所有标签以向量形式赋值给wd
wd<-names(Bio_list)
#统计每个词汇的词频,并以向量形式给num
num<-NULL
for (j in 1:length(Bio_list)) {
num<-c(num,length(Bio_list[[j]]))
}
#将词汇和词频组成数据框,用于后期可视化
Paper_wd<-data.frame(a=wd,b=num)
#建立一个新的向量,储存and in the等这些定冠词、连词等英文中无实意的单词,用于过滤数据框中这部分数据
words<-c("and","in","the","of","on","a")
#新定义Paper_wd2数据框用于储存过滤了无实意英文单词的文献词汇及词频数据框
Paper_wd2<-data.frame()
for (i in 1:nrow(Paper_wd)) {
if(as.character(Paper_wd[i,1]) %in% words){next}
else{Paper_wd2<-rbind(Paper_wd2,Paper_wd[i,])}
}
#加载词云图函数包,用于可视化最新发表的文献题目中的词汇
library(wordcloud2)
wordcloud2(Paper_wd2)
从词云图可以看出新发表文献主要还是以研究分析、基因、表达等内容较多。当然还有The这些首字母大写的定冠词没有过滤掉。
本次重点,掌握自学R语言函数包的技能,强化R语言编程能力。掌握R语言爬虫的三个重要函数read_html、html_nodes、html_text使用方法及搭配管道符使用的技能。了解HTML语言本次涉及到的可视化内容我将在下一讲专门介绍R语言可视化知识。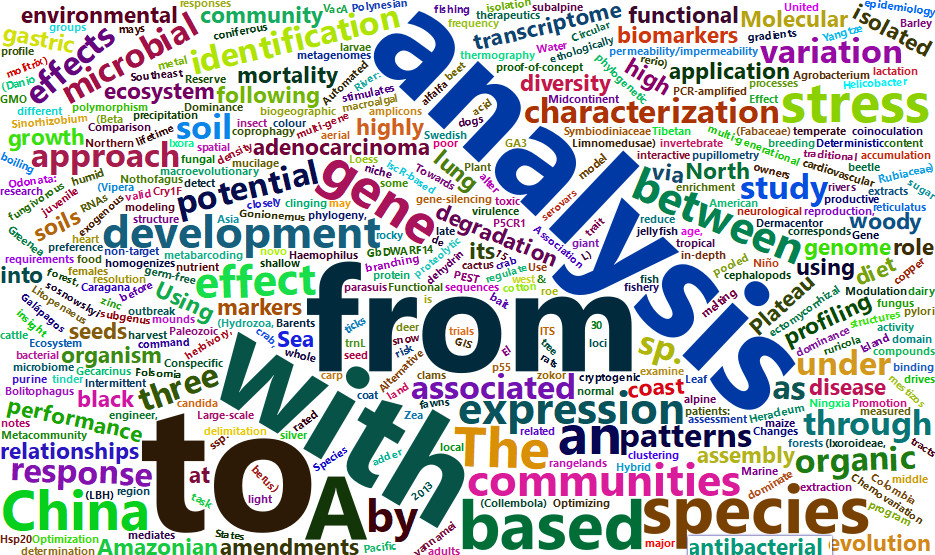
往期精彩文章回顾
从零开始入门R语言编程
RNA-seq中的那些统计学问题(一)为什么是负二项分布




